5 Loops en R. Funciones base de R
(‘“Looping”, “cycling”, “iterating” or just replicating instructions’)
Trabajaremos con conjunto de datos llamada iris, la base tiene información sobre el largo y ancho de sépalos y pétalos de 50 flores de tres especies de plantas.
5.1 apply()
Aplicas instrucciones a filas o columnas de un data.frame o matriz.
Si queremos estimar la media de cada una de mis cuatro variables de la tabla iris entonces podría usar lo siguiente:
mean(iris$Sepal.Length)
#> [1] 5.843333
mean(iris$Sepal.Width)
#> [1] 3.057333
mean(iris$Petal.Length)
#> [1] 3.758
mean(iris$Petal.Width)
#> [1] 1.199333o
c(mean(iris$Sepal.Length), mean(iris$Sepal.Width), mean(iris$Petal.Length), mean(iris$Petal.Width))
#> [1] 5.843333 3.057333 3.758000 1.199333oo
colMeans(iris[,1:4])
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 5.843333 3.057333 3.758000 1.199333Pero podríamos usar apply()
apply(iris, 2, mean)
#> Warning in mean.default(newX[, i], ...): argument is not
#> numeric or logical: returning NA
#> Warning in mean.default(newX[, i], ...): argument is not
#> numeric or logical: returning NA
#> Warning in mean.default(newX[, i], ...): argument is not
#> numeric or logical: returning NA
#> Warning in mean.default(newX[, i], ...): argument is not
#> numeric or logical: returning NA
#> Warning in mean.default(newX[, i], ...): argument is not
#> numeric or logical: returning NA
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> NA NA NA NA
#> Species
#> NA¿Porqué sale error o NA?
Para ir tomando ritmo resolvamos los siguientes ejercicios:
1) Ahora quiero la raíz cuadrada de la media que previamente es elevada a la 5ta potencia
apply(iris[,1:4], 2, function(x){
x.1 <- sqrt(mean(x)^5)
})
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 82.537565 16.343948 27.377383 1.5752512) El resultado de la raíz cuadrada la quiero multiplicar por 15 y dividir entre 2
3) Si el resultado es mayor a 1000 que devuelva un 0
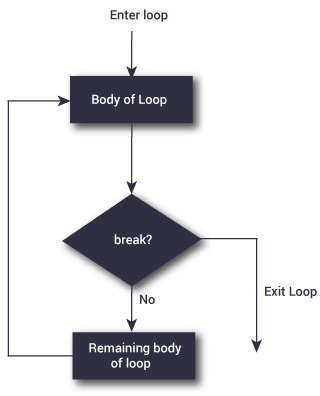
5.2 for()
Sirve para repetir n veces una o más instrucciones. La estructura es:
En el siguiente ejemplo usaremos for() para obtener un boxplot para cada variable de pétalo y sépalo.
Solución 1.
par(mfrow=c(2,2))
boxplot(iris$Sepal.Length ~ iris$Species, ylab = "Sepal.Length",
xlab = "Specie")
boxplot(iris$Sepal.Width ~ iris$Species, ylab = "Sepal.Width",
xlab = "Specie")
boxplot(iris$Petal.Length ~ iris$Species, ylab = "Petal.Length",
xlab = "Specie")
boxplot(iris$Petal.Width ~ iris$Species, ylab = "Petal.Width",
xlab = "Specie")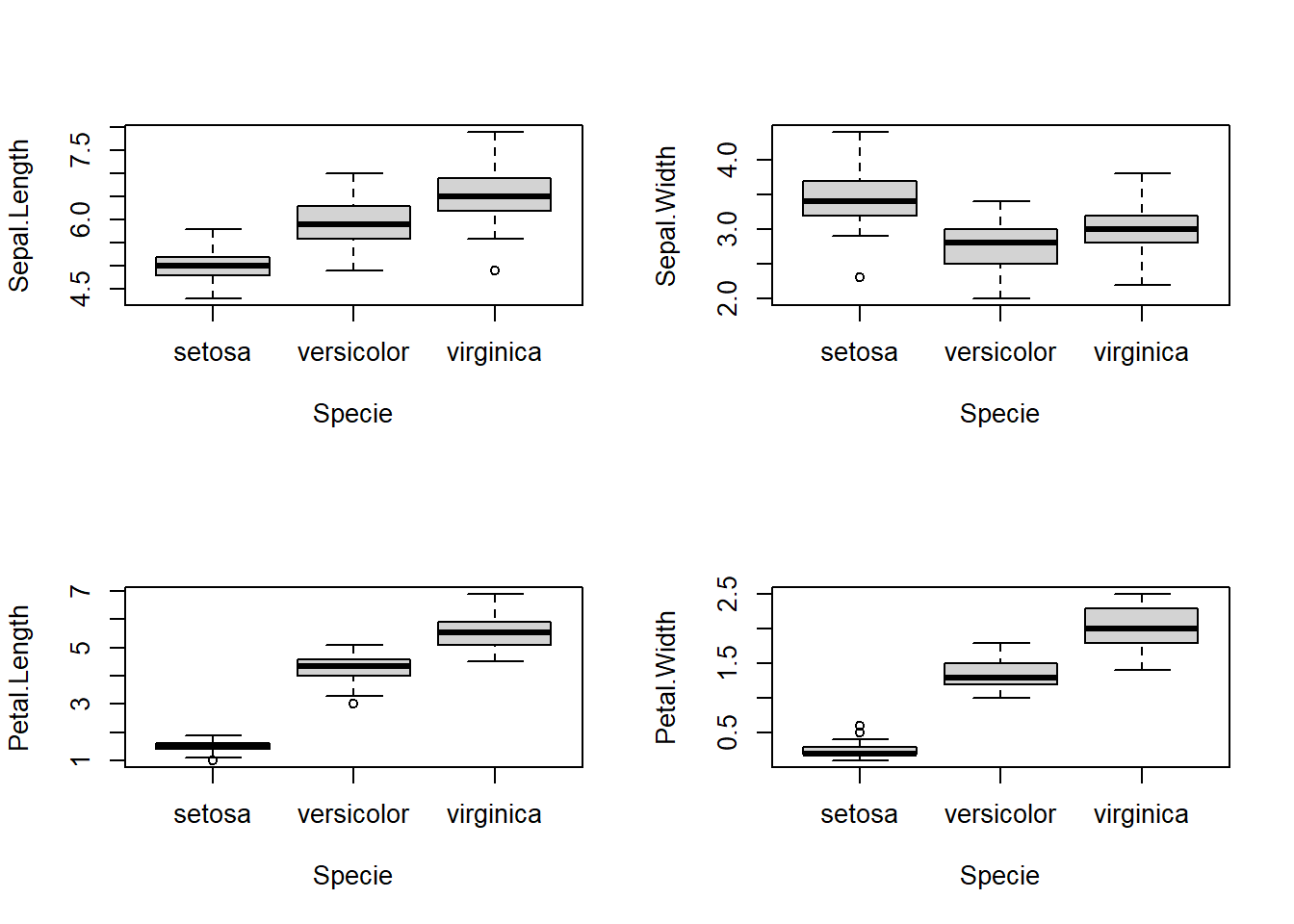
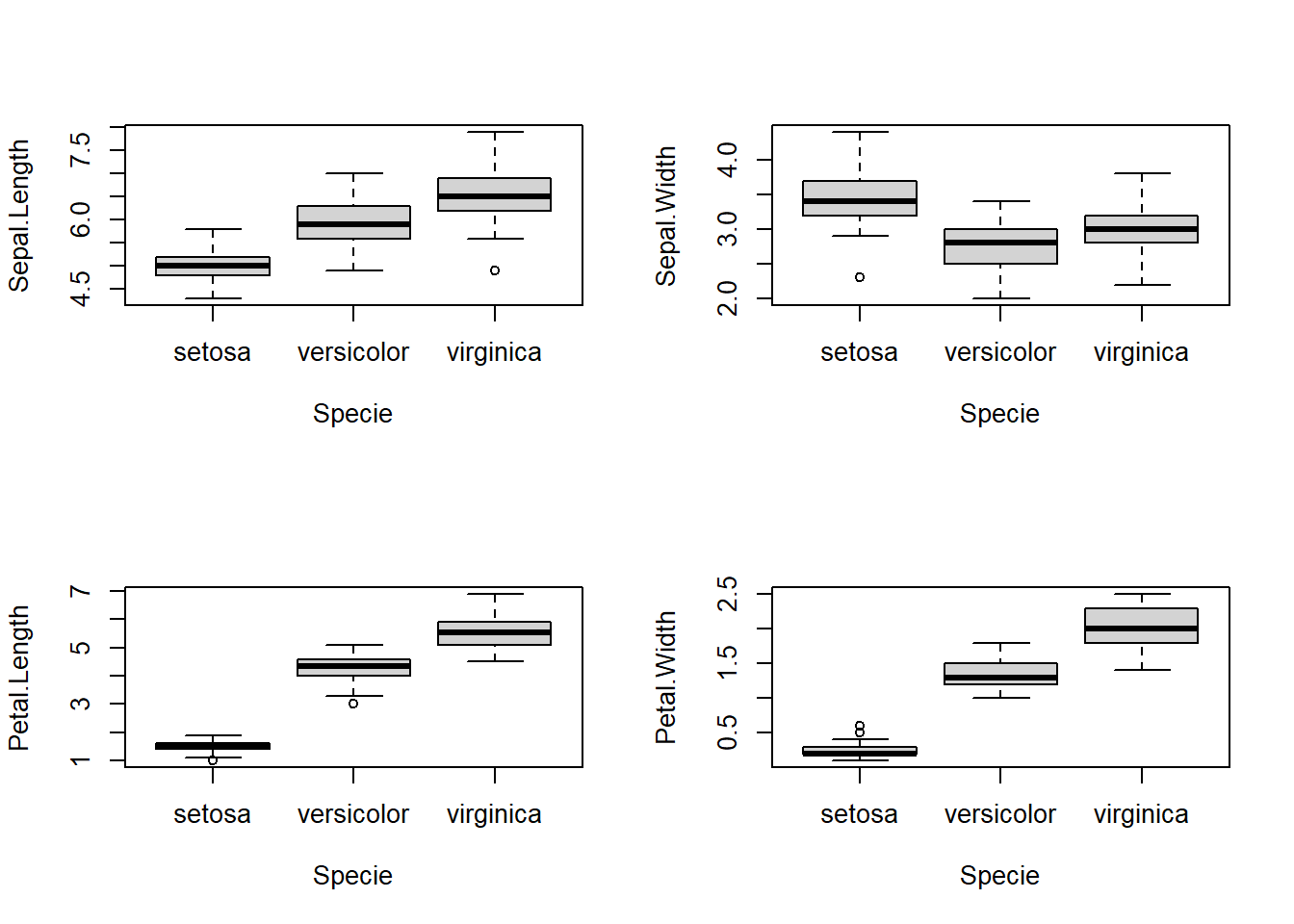
Solución 2.
par(mfrow=c(2,2))
for(i in 1:4){
boxplot(iris[[i]] ~ iris$Species, ylab = names(iris)[i], xlab = "Specie")
}
Además, podemos crear vectores, listas o data.frames, pero previamente debemos crear un objeto que va a almacenar nuestra salida de cada iteración (repetición de instrucciones) y que debe tener las propiedades que queremos de salida.
Vector
prueba <- vector()
for(i in 1:4){
prueba[[i]] <- mean(iris[[i]])
}
prueba
#> [1] 5.843333 3.057333 3.758000 1.199333Data.frame
prueba <- data.frame(Especie = "",
"MSepal.Length" = 0,
"SDSepal.Length" = 0)
prueba
#> Especie MSepal.Length SDSepal.Length
#> 1 0 0
# c("setosa", "versicolor", "virginica") = unique(iris$Species)
for(i in 1:3){
i.1 <- iris[iris$Species == unique(iris$Species)[[i]],]
prueba[i,] <- data.frame(Especie = as.character(unique(iris$Species)[[i]]),
"MSepal.Length" = mean(i.1$Sepal.Length),
"SDSepal.Length" = sd(i.1$Sepal.Length))
}
prueba
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#> 2 versicolor 5.936 0.5161711
#> 3 virginica 6.588 0.6358796Lista
prueba <- list()
# c("setosa", "versicolor", "virginica") = unique(iris$Species)
for(i in 1:3){
i.1 <- iris[iris$Species == unique(iris$Species)[[i]],]
prueba[[i]] <- data.frame(Especie = as.character(unique(iris$Species)[[i]]),
"MSepal.Length" = mean(i.1$Sepal.Length),
"SDSepal.Length" = sd(i.1$Sepal.Length))
}
prueba
#> [[1]]
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#>
#> [[2]]
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> [[3]]
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796Para colapsar la lista en un unico objeto podemos usar la función do.call()
prueba <- do.call(rbind, prueba)
prueba
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#> 2 versicolor 5.936 0.5161711
#> 3 virginica 6.588 0.63587965.3 sapply()
sapply trabaja con vectores o listas y las salidas usuales pueden ser matrices, con varios array, una lista o un vector, es decir, puede hacer de todo!
Veamos algunas de sus aplicaciones más comunes.
1. Obtener un vector numérico. Estimar la media para cada variable del conjunto de datos iris.
2. Obtener una matriz con arrays que puedes transformar a data.frame. Ojo el resultado de iteración lo agrega en una “columna”.
sapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> [,1] [,2]
#> Especie "setosa" "versicolor"
#> MSepal.Length "5.006" "5.936"
#> SDSepal.Length "0.352489687213451" "0.516171147063863"
#> [,3]
#> Especie "virginica"
#> MSepal.Length "6.588"
#> SDSepal.Length "0.635879593274432"Si quiero tener una salida más amigable como un data.frame tendríamos que usar otras funciones, por ejemplo:
library(magrittr)# para usar %>%
sapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
}) %>% as.data.frame() %>% t() %>% as.data.frame()
#> Especie MSepal.Length SDSepal.Length
#> V1 setosa 5.006 0.352489687213451
#> V2 versicolor 5.936 0.516171147063863
#> V3 virginica 6.588 0.6358795932744323. Obtener una lista. Se necesita que las salidas tengan dimensiones distintas, de lo contrario lo juntara en una matriz.
sapply(3:9, seq)
#> [[1]]
#> [1] 1 2 3
#>
#> [[2]]
#> [1] 1 2 3 4
#>
#> [[3]]
#> [1] 1 2 3 4 5
#>
#> [[4]]
#> [1] 1 2 3 4 5 6
#>
#> [[5]]
#> [1] 1 2 3 4 5 6 7
#>
#> [[6]]
#> [1] 1 2 3 4 5 6 7 8
#>
#> [[7]]
#> [1] 1 2 3 4 5 6 7 8 9
sapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
if(unique(x.1$Species) == "setosa"){
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length))
} else {
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
}
return(x.1)
})
#> [[1]]
#> Especie MSepal.Length
#> "setosa" "5.006"
#>
#> [[2]]
#> Especie MSepal.Length SDSepal.Length
#> "versicolor" "5.936" "0.516171147063863"
#>
#> [[3]]
#> Especie MSepal.Length SDSepal.Length
#> "virginica" "6.588" "0.635879593274432"5.4 tapply()
La función tapply() aplica una función a un vector x y agrupa la salida de acuerdo con un vector y del mismo largo. El vector y es categórico e idealmente debe tener distintos niveles de factor.
Ejemplo.
tapply(iris$Sepal.Width, iris$Species, mean)
#> setosa versicolor virginica
#> 3.428 2.770 2.9745.5 mapply()
La función mapply() puede ser usada bajo la misma logica de sapply pero usando más de un vector. Los vectores iteractuan por posición, es decir, el primer elemento del vector y y el vector 2, seguido del segundo elemento del vector 1 y vector 2, etc.
Ejemplos:
mapply(mean, iris$Sepal.Length, iris$Petal.Length)
#> [1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8
#> [14] 4.3 5.8 5.7 5.4 5.1 5.7 5.1 5.4 5.1 4.6 5.1 4.8 5.0
#> [27] 5.0 5.2 5.2 4.7 4.8 5.4 5.2 5.5 4.9 5.0 5.5 4.9 4.4
#> [40] 5.1 5.0 4.5 4.4 5.0 5.1 4.8 5.1 4.6 5.3 5.0 7.0 6.4
#> [53] 6.9 5.5 6.5 5.7 6.3 4.9 6.6 5.2 5.0 5.9 6.0 6.1 5.6
#> [66] 6.7 5.6 5.8 6.2 5.6 5.9 6.1 6.3 6.1 6.4 6.6 6.8 6.7
#> [79] 6.0 5.7 5.5 5.5 5.8 6.0 5.4 6.0 6.7 6.3 5.6 5.5 5.5
#> [92] 6.1 5.8 5.0 5.6 5.7 5.7 6.2 5.1 5.7 6.3 5.8 7.1 6.3
#> [105] 6.5 7.6 4.9 7.3 6.7 7.2 6.5 6.4 6.8 5.7 5.8 6.4 6.5
#> [118] 7.7 7.7 6.0 6.9 5.6 7.7 6.3 6.7 7.2 6.2 6.1 6.4 7.2
#> [131] 7.4 7.9 6.4 6.3 6.1 7.7 6.3 6.4 6.0 6.9 6.7 6.9 5.8
#> [144] 6.8 6.7 6.7 6.3 6.5 6.2 5.9
mapply(sum, iris$Sepal.Length, iris$Petal.Length, iris$Sepal.Width)
#> [1] 10.0 9.3 9.2 9.2 10.0 11.0 9.4 9.9 8.7 9.5 10.6
#> [12] 9.8 9.2 8.4 11.0 11.6 10.6 10.0 11.2 10.4 10.5 10.3
#> [23] 9.2 10.1 10.1 9.6 10.0 10.2 10.0 9.5 9.5 10.3 10.8
#> [34] 11.1 9.5 9.4 10.3 9.9 8.7 10.0 9.8 8.1 8.9 10.1
#> [45] 10.8 9.2 10.5 9.2 10.5 9.7 14.9 14.1 14.9 11.8 13.9
#> [56] 13.0 14.3 10.6 14.1 11.8 10.5 13.1 12.2 13.7 12.1 14.2
#> [67] 13.1 12.6 12.9 12.0 13.9 12.9 13.7 13.6 13.6 14.0 14.4
#> [78] 14.7 13.4 11.8 11.7 11.6 12.4 13.8 12.9 13.9 14.5 13.0
#> [89] 12.7 12.0 12.5 13.7 12.4 10.6 12.5 12.9 12.8 13.4 10.6
#> [100] 12.6 15.6 13.6 16.0 14.8 15.3 17.2 11.9 16.5 15.0 16.9
#> [111] 14.8 14.4 15.3 13.2 13.7 14.9 15.0 18.2 17.2 13.2 15.8
#> [122] 13.3 17.2 13.9 15.7 16.4 13.8 14.0 14.8 16.0 16.3 18.1
#> [133] 14.8 14.2 14.3 16.8 15.3 15.0 13.8 15.4 15.4 15.1 13.6
#> [144] 15.9 15.7 14.9 13.8 14.7 15.0 14.0O podría hacer una función:
f <- function(a,b,c) a+b/c
mapply(f, iris$Sepal.Length, iris$Petal.Length, iris$Sepal.Width)
#> [1] 5.500000 5.366667 5.106250 5.083871 5.388889
#> [6] 5.835897 5.011765 5.441176 4.882759 5.383871
#> [11] 5.805405 5.270588 5.266667 4.666667 6.100000
#> [16] 6.040909 5.733333 5.500000 6.147368 5.494737
#> [21] 5.900000 5.505405 4.877778 5.615152 5.358824
#> [26] 5.533333 5.470588 5.628571 5.611765 5.200000
#> [31] 5.316129 5.841176 5.565854 5.833333 5.383871
#> [36] 5.375000 5.871429 5.288889 4.833333 5.541176
#> [41] 5.371429 5.065217 4.806250 5.457143 5.600000
#> [46] 5.266667 5.521053 5.037500 5.705405 5.424242
#> [51] 8.468750 7.806250 8.480645 7.239130 8.142857
#> [56] 7.307143 7.724242 6.275000 8.186207 6.644444
#> [61] 6.750000 7.300000 7.818182 7.720690 6.841379
#> [66] 8.119355 7.100000 7.318519 8.245455 7.160000
#> [71] 7.400000 7.528571 8.260000 7.778571 7.882759
#> [76] 8.066667 8.514286 8.366667 7.551724 7.046154
#> [81] 7.083333 7.041667 7.244444 7.888889 6.900000
#> [86] 7.323529 8.216129 8.213043 6.966667 7.100000
#> [91] 7.192308 7.633333 7.338462 6.434783 7.155556
#> [96] 7.100000 7.148276 7.682759 6.300000 7.164286
#> [101] 8.118182 7.688889 9.066667 8.231034 8.433333
#> [106] 9.800000 6.700000 9.472414 9.020000 8.894444
#> [111] 8.093750 8.362963 8.633333 7.700000 7.621429
#> [116] 8.056250 8.333333 9.463158 10.353846 8.272727
#> [121] 8.681250 7.350000 10.092857 8.114815 8.427273
#> [126] 9.075000 7.914286 7.733333 8.400000 9.133333
#> [131] 9.578571 9.584211 8.400000 8.121429 8.253846
#> [136] 9.733333 7.947059 8.174194 7.600000 8.641935
#> [141] 8.506452 8.545161 7.688889 8.643750 8.427273
#> [146] 8.433333 8.300000 8.233333 7.788235 7.6000005.6 lapply()
La función lapply es una de las funciones más usadas en R, permite realizar iteraciones sobre una lista, array o vector y devuelve siempre una lista. Esto tiene varias ventajas, por ejemplo, que puedes guardar cualquier tipo clase de R.
Vector
lapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> [[1]]
#> Especie MSepal.Length SDSepal.Length
#> "setosa" "5.006" "0.352489687213451"
#>
#> [[2]]
#> Especie MSepal.Length SDSepal.Length
#> "versicolor" "5.936" "0.516171147063863"
#>
#> [[3]]
#> Especie MSepal.Length SDSepal.Length
#> "virginica" "6.588" "0.635879593274432"Data.frame
lapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- data.frame("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> [[1]]
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#>
#> [[2]]
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> [[3]]
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796Para colapsar la lista en un unico data.frame podemos usar la función do.call()
lapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- data.frame("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
}) %>% do.call(rbind, .)
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#> 2 versicolor 5.936 0.5161711
#> 3 virginica 6.588 0.6358796lista
lapply(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- list("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> [[1]]
#> [[1]]$Especie
#> [1] "setosa"
#>
#> [[1]]$MSepal.Length
#> [1] 5.006
#>
#> [[1]]$SDSepal.Length
#> [1] 0.3524897
#>
#>
#> [[2]]
#> [[2]]$Especie
#> [1] "versicolor"
#>
#> [[2]]$MSepal.Length
#> [1] 5.936
#>
#> [[2]]$SDSepal.Length
#> [1] 0.5161711
#>
#>
#> [[3]]
#> [[3]]$Especie
#> [1] "virginica"
#>
#> [[3]]$MSepal.Length
#> [1] 6.588
#>
#> [[3]]$SDSepal.Length
#> [1] 0.6358796