7 Loops en R. Paralelizar
Existen diversas estrategias de paralelización y paquetes que ayudan a aplicarlos en R (e.g., parallel, foreach). En esta sección usaremos el paquete future que trata de simplificar la aplicación de la paralelización en R para objetos de distintas clases. El paquete future tiene diversas estrategias de paralelización que pueden interactuar con la mayoría de las funciones y paquetes de R.

Entre las estrategias principales se encuentran las siguientes:

Las dos estrategias más populares son multicore y multiprocess, sin embargo, multicore solo está disponible para Linux.
Secuencial
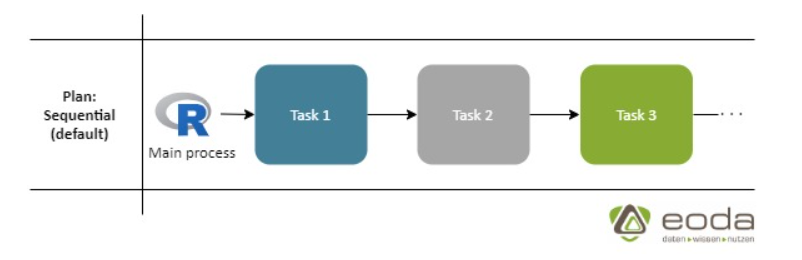
Multisession vs Multicore
7.1 Multisession
Paso 0. Instalar y abrir el paquete future
#>
#> Attaching package: 'magrittr'
#> The following object is masked from 'package:purrr':
#>
#> set_names
install.packages("future", dependencies = TRUE)
library(future)Paso 1. Determinar cuántos cores o workers están disponibles, en este ejemplo usaremos la función nativa del paquete future llamada availableCores():
availableCores()
#> system
#> 8
availableCores()/2
#> system
#> 4
availableCores()-2
#> system
#> 6Paso 2. Correr la estrategia multisesssion usando la función plan()
plan(strategy = multisession, gc = TRUE, workers = 4)Paso 3. Cerrar el multisession
Opción 1
plan(sequential)Opción 2
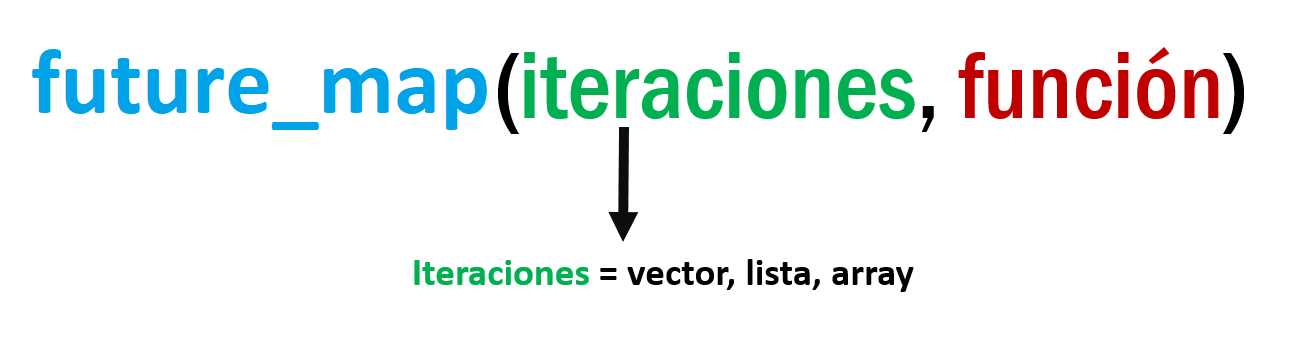
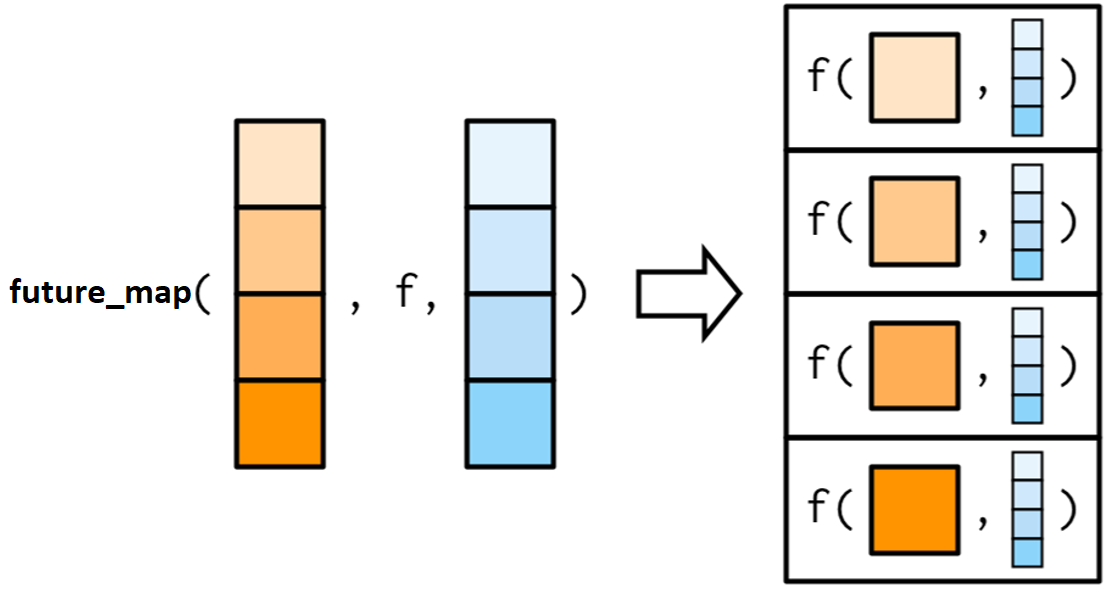
7.3 future_map()
La función future_map es el simil de map. La función permite realizar iteraciones sobre una lista, array o vector y devuelve siempre una lista. Esto tiene varias ventajas, por ejemplo, que puedes guardar cualquier clase de R.
Abrimos future y furrr, y escogemos una estrategia de paralelización
plan(strategy = multisession, gc = TRUE, workers = 4)Vector como iteración
entrada <- 1:3
future_map(entrada, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> [[1]]
#> Especie MSepal.Length SDSepal.Length
#> "setosa" "5.006" "0.352489687213451"
#>
#> [[2]]
#> Especie MSepal.Length SDSepal.Length
#> "versicolor" "5.936" "0.516171147063863"
#>
#> [[3]]
#> Especie MSepal.Length SDSepal.Length
#> "virginica" "6.588" "0.635879593274432"lista como iteración
Imaginemos que previamente hice un proceso donde separé las filas de cada especie y las guardé por separado en una lista.
entrada <- iris %>% split(iris$Species)
names(entrada)
#> [1] "setosa" "versicolor" "virginica"
class(entrada)
#> [1] "list"Ahora la lista tiene tres data.frames con los datos de cada especie y la usaremos como entrada en la función future_map(). La lista tiene 3 elementos por lo que hará 3 iteraciones. En la iteración 1 tomará el primer data.frame de la lista, en la iteración 2 el segundo data.frame de la lista y en la iteración 3 tomará el tercer data.frame de la lista.
ejemplo <- future_map(entrada, function(x){
x.1 <- data.frame("Especie" = as.character(unique(x$Species)),
"MSepal.Length" = mean(x$Sepal.Length),
"SDSepal.Length" = sd(x$Sepal.Length))
return(x.1)
})
ejemplo
#> $setosa
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#>
#> $versicolor
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> $virginica
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796Colapsar la lista
Ya que la lista de salida tiene data.frames con columnas con el mismo nombre podemos colapsarlos en un único data.frame usando la función do.call() y rbind(), la ultima indica de que forma se puede colapsar la lista en este caso rbind indica que las apile por filas, de tal forma que las columnas se mantienen integras y lo único que incrementa son las filas.
Antes es importante quitar los NULL. En ocasiones cuando algo no ocurre como deseamos en lugar de dejar que falle el loop guardamos el resultado como un NULL, así sabemos que los NULL dentro de nuestra lista son errores. Por ejemplo, supongamos que no queremos tener información de setosa porque sabemos que la información es incorrecta, así que aplicaremos una iteración y cuando lleguemos a esa especie devolverá un NULL.
ejemplo <- future_map(entrada, function(x){
if(unique(x$Species) == "setosa"){
x.1 <- NULL
} else {
x.1 <- data.frame("Especie" = as.character(unique(x$Species)),
"MSepal.Length" = mean(x$Sepal.Length),
"SDSepal.Length" = sd(x$Sepal.Length))
}
return(x.1)
})
ejemplo
#> $setosa
#> NULL
#>
#> $versicolor
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> $virginica
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796Ahora antes de hacer un do.call() necesitamos quitar el elemento NULL de nuestra lista. En este ejemplo es sencillo porque tenemos solo tres elementos, pero imaginemos su importancia cuando tengamos docenas, cientos o miles de elementos en nuestra lista.
Para quitar los NULL podemos recurrir a las funciones base de R:
Filter(Negate(is.null), ejemplo)
#> $versicolor
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> $virginica
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796O podemos usar la función compact de purrr
purrr::compact(ejemplo)
#> $versicolor
#> Especie MSepal.Length SDSepal.Length
#> 1 versicolor 5.936 0.5161711
#>
#> $virginica
#> Especie MSepal.Length SDSepal.Length
#> 1 virginica 6.588 0.6358796
ejemplo <- purrr::compact(ejemplo)
do.call(rbind, ejemplo)
#> Especie MSepal.Length SDSepal.Length
#> versicolor versicolor 5.936 0.5161711
#> virginica virginica 6.588 0.6358796También podríamos colapsar la lista por columnas usando la función cbind, de tal forma que la única fila que tenemos se queda integra y lo que cambia es el no. de columnas.
do.call(cbind, ejemplo)
#> versicolor.Especie versicolor.MSepal.Length
#> 1 versicolor 5.936
#> versicolor.SDSepal.Length virginica.Especie
#> 1 0.5161711 virginica
#> virginica.MSepal.Length virginica.SDSepal.Length
#> 1 6.588 0.6358796Cerrar la paralelización
7.4 map_dfr y map_dfc
Para ahorrarnos el paso de usar la función do.call() para colapsar las listas en un data.frame podemos usar las funciones future_map_dfr() y future_map_dfc().
Es igual a la función future_map (i.e., tiene la misma estructura y trabaja con vectores, listas, arrays) pero la salida siempre es un data.frame lo que implica que en las instrucciones que aplicas en cada iteración el resultado siempre debe ser un data.frame. Si la salida es un vector numérico, un vector de caracteres, una lista, array u otra clase entonces te marcará un error.
Abrimos future y furrr, y escogemos una estrategia de paralelización
plan(strategy = multisession, gc = TRUE, workers = 4)7.4.0.1 future_map_dfr()
En nuestro ejemplo anterior, esta función sustituye el do.call(rbind, ejemplo). Nota. Dejaremos setosa.
Usando una lista
future_map_dfr(entrada, function(x){
x.1 <- data.frame("Especie" = as.character(unique(x$Species)),
"MSepal.Length" = mean(x$Sepal.Length),
"SDSepal.Length" = sd(x$Sepal.Length))
return(x.1)
})
#> Especie MSepal.Length SDSepal.Length
#> 1 setosa 5.006 0.3524897
#> 2 versicolor 5.936 0.5161711
#> 3 virginica 6.588 0.6358796Usando un vector
future_map_dfr(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> # A tibble: 3 x 3
#> Especie MSepal.Length SDSepal.Length
#> <chr> <chr> <chr>
#> 1 setosa 5.006 0.352489687213451
#> 2 versicolor 5.936 0.516171147063863
#> 3 virginica 6.588 0.6358795932744327.4.1 future_map_dfc()
En nuestro ejemplo anterior, esta función sustituye el do.call(cbind, ejemplo).
Usando una lista
future_map_dfc(entrada, function(x){
x.1 <- data.frame("Especie" = as.character(unique(x$Species)),
"MSepal.Length" = mean(x$Sepal.Length),
"SDSepal.Length" = sd(x$Sepal.Length))
return(x.1)
})
#> New names:
#> * `Especie` -> `Especie...1`
#> * `MSepal.Length` -> `MSepal.Length...2`
#> * `SDSepal.Length` -> `SDSepal.Length...3`
#> * `Especie` -> `Especie...4`
#> * `MSepal.Length` -> `MSepal.Length...5`
#> * `SDSepal.Length` -> `SDSepal.Length...6`
#> * `Especie` -> `Especie...7`
#> * `MSepal.Length` -> `MSepal.Length...8`
#> * `SDSepal.Length` -> `SDSepal.Length...9`
#> Especie...1 MSepal.Length...2 SDSepal.Length...3
#> 1 setosa 5.006 0.3524897
#> Especie...4 MSepal.Length...5 SDSepal.Length...6
#> 1 versicolor 5.936 0.5161711
#> Especie...7 MSepal.Length...8 SDSepal.Length...9
#> 1 virginica 6.588 0.6358796Usando un vector
future_map_dfc(1:3, function(x){
x.1 <- iris[iris$Species == unique(iris$Species)[[x]],]
x.1 <- c("Especie" = as.character(unique(iris$Species)[[x]]),
"MSepal.Length" = mean(x.1$Sepal.Length),
"SDSepal.Length" = sd(x.1$Sepal.Length))
return(x.1)
})
#> New names:
#> * `` -> `...1`
#> * `` -> `...2`
#> * `` -> `...3`
#> # A tibble: 3 x 3
#> ...1 ...2 ...3
#> <chr> <chr> <chr>
#> 1 setosa versicolor virginica
#> 2 5.006 5.936 6.588
#> 3 0.352489687213451 0.516171147063863 0.635879593274432¿Y si tenemos NULL de salida o un error en el proceso?
7.5 future_map_dbl,future_ map_chr y future_map_lgl
Este grupo de funciones realiza iteraciones sobre una lista, array o vector y devuelve siempre un vector numérico (map_dbl), carácter (map_chr) o lógico (map_lgl).
7.5.1 future_map_dbl()
future_map_dbl(1:4, function(x){
x.1 <- sqrt(mean(iris[[x]])^5)
return(x.1)
})
#> [1] 82.537565 16.343948 27.377383 1.5752517.5.2 future_map_chr()
future_map_chr(entrada, function(x){
x.1 <- unique(as.character(unique(x$Species)))
return(x.1)
})
#> setosa versicolor virginica
#> "setosa" "versicolor" "virginica"7.5.3 future_map_lgl()
future_map_lgl(entrada, function(x){
x.1 <- unique(as.character(unique(x$Species))) == "setosa"
return(x.1)
})
#> setosa versicolor virginica
#> TRUE FALSE FALSE7.6 Incluir una condicional para detectar posibles errores
prueba <- tryCatch(future_map_lgl(entrada, function(x){
x.1 <- unique(as.character(unique(x$Species))) == "setosa"
if(inherits(x.1, "error")){
# Si existe un error primero cierro los sub procesos
a <- ps::ps()
a <- a[which(a$name =="Rscript.exe"),1]
for(i in a){
tools::pskill(i)
}
#Despues detengo el loop
stop("oh error")
}
return(x.1)
}), error = function(err)err)
if(inherits(prueba, "error")){
#Si es parte de una función sirve guardar un posible error con tryCatch
# Si existe un error primero cierro los sub procesos
a <- ps::ps()
a <- a[which(a$name =="Rscript.exe"),1]
for(i in a){
tools::pskill(i)
}
#Despues detengo el loop
stop("oh error")
}Cerrar la paralelización